博客
用 AlphaFold 和 RoseTTAFold 进行蛋白质结构预测和药物发现
对于蛋白质来说,结构驱动功能。蛋白质通过一个复杂的折叠过程达到其功能形式。作为生物体内四大生物大分子中最丰富的一种,了解生命意味着需要了解蛋白质。
正如它们的结构和功能驱动着生物体的进程一样,错误的蛋白质结构在错误的地方会导致疾病。为了减轻症状,有时甚至治愈疾病,意味着要了解和干预变异的蛋白质。对于许多疾病,无论是由病毒、细菌病原体还是我们自己的异常蛋白质引起的,治疗疾病意味着针对相关蛋白质。
青霉素是世界上最著名的抗生素,它通过结合并抑制细菌用来构建由肽聚糖组成的细胞壁的蛋白酶而杀死细菌。虽然众所周知青霉素是偶然发现的,但现代药物开发现在有了工具,可以有意识地设计药物来治疗疾病。
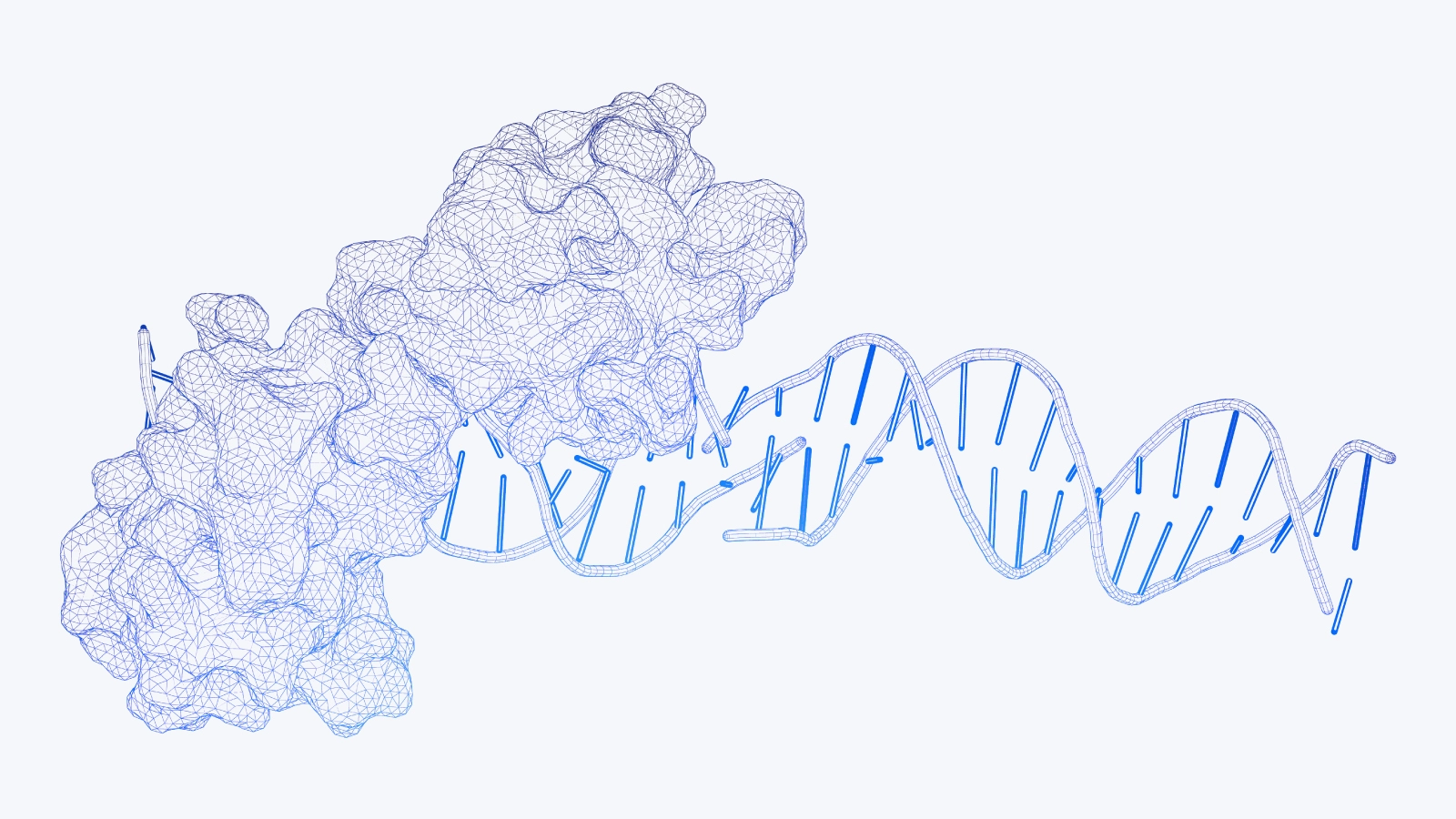
现代深度学习技术最近对科学领域几十年来的巨大挑战做出了令人难以置信的贡献:仅根据氨基酸序列预测蛋白质的结构。根据一些估计,在 2020 年举行的第 14 届蛋白质结构预测关键评估会议(CASP14)上,DeepMind 的 AlphaFold 第二次迭代基本解决了蛋白质结构预测问题。AlphaFold 利用了当代自然语言处理的技术与基于注意力的转化器模型,以及传统的生物信息学方法来准确预测蛋白质的结构,没有其他算法能更加接近。
AlphaFold 和受其发展启发的类似模型导致了结构生物学的快速进展。毫不奇怪,许多人认为这一突破是开发治疗以前无法治愈的疾病的一种手段。在这篇文章中,我们将讨论蛋白质结构预测问题的背景,以及引入可获得的、准确的蛋白质结构估计对药物开发意味着什么。我们还将介绍到目前为止所做的一些工作,并讨论如何开始使用 AlphaFold 和 RoseTTAFold 模型,包括公开的在线资源以及硬件和软件的考虑。
问题的范围
地球上的每一个生物都依赖蛋白质来运作。即使是技术上不被认为是有生命的病毒,也完全依赖宿主细胞产生的蛋白质进行繁殖。
与脂质、核酸和碳水化合物一样,蛋白质是四大生物大分子之一。蛋白质具有多种作用,包括产生力量和运动(如肌肉蛋白肌球)、结构(如构成细胞骨架的微管)、计算(如神经信号中调节动作电位的蛋白质),以及作为化学催化剂(任何数量的蛋白酶)。
考虑到它们是由一维的氨基酸序列构成的,使蛋白质变得极为迷人。复杂的蛋白质复合物的折叠就像用一组弦来建造一个喷气式发动机,使用的珠子图案恰到好处。

为什么要解决蛋白质折叠的问题?
蛋白质结构的知识告诉我们它们是如何在健康和疾病中运作的,了解蛋白质结构可以揭示出改变疾病行为和开发药物来解决最困难的病症的见解。
蛋白质结构数据库也在增长,但通过冷冻电镜和 X 射线晶体学评估特定蛋白质的结构是一个具有挑战性的过程,可能需要几年时间。
直到最近,通过计算从序列上预测一个准确的蛋白质结构并不容易,而且通常也不是很准确。为了理解这个原因,考虑问题的规模是有帮助的。
蛋白质的大小不一,氨基酸的聚合体从一个小肽到有资格被归类为蛋白质的边界也不一样。大多数蛋白质都要小得多:平均有几百个氨基酸残基,但也可以跨越到几千个。最大的已知蛋白质是 Titin,长度从 27,000 到约 35,000 个氨基酸不等。
我们可以搜索一个给定的氨基酸序列的所有可能的构象,并保留最有利的构象,对吗?这样做可能需要的时间比宇宙的年龄还长。一个只有 100 个氨基酸残基的蛋白质将有 3198 个或不可估量的 1059 个不同结构需要考虑。如果把这一点应用到钛蛋白上,科学发现将处于停滞状态。
在药物开发中使用人工智能模型
AlphaFold 在提高医学和保健方面的潜力是一个革命性的进步。尽管个性化医疗和个人基因组测序的进展仍在发展之中,但 AlphaFold 提供准确的蛋白质结构预测的能力为推进药物设计带来了巨大的希望。目前已经有许多令人兴奋的项目正在探索其众多的应用。
2021 年,在发表 AlphaFold2 论文大约一周后,DeepMind 宣布了 AlphaFold 蛋白质结构数据库。该数据库将人类蛋白质组(人类基因组中编码的所有蛋白质集合)的蛋白质结构覆盖率从约 17% 扩大到氨基酸残基水平的 98% 以上。
几个月后,DeepMind 扩大了覆盖范围,包括来自另外 21 种模式生物的 36 万个蛋白质结构预测。在发布的同时,还对数据库的程序化访问进行了一些改进。
该数据库是公开的,这是消除使用 AlphaFold 结构的障碍的重要一步,也是对向全世界推广其工具的重大贡献。用 AlphaFold 或 RoseTTAFold 模型预测结构的端到端管道有很大的硬件要求、能源成本和运行时间。
这两个模型都需要一个高性能的计算机,有大量的存储空间,多个快速的 CPU 核心,大量的内存用于对准过程中的序列搜索,以及一个多 GPU 配置,有充足的内存和计算能力用于结构预测。
公共数据库通过减少不同方面重复生成相同结构的需要来减少能源的浪费。这也使得研究实验室和药品开发商更容易利用这些结构,即使他们没有自己的计算资源来生成结构。
AlphaFold 蛋白质数据库促进了基于预测结构的小分子疗法的发展,但是仍然需要生成高质量的结构预测来设计蛋白质疗法,并探索多种可能的构象状态。
发现一种新的 CDK20 激酶抑制剂
通过将 AlphaFold 整合到一个预先存在的目标识别和小分子生成管道中,Ren 等人证明了在生成 "命中 "方面的效率提高,即能够与目标疾病所涉及的蛋白质结合的小分子。命中率的生成和验证只是利用现代技术开发新药的漫长道路上的前几个步骤。
Ren 和他的同事使用了一个市售的软件包 PandaOmics,将细胞周期蛋白依赖性激酶 20(CDK20)确定为他们感兴趣的疾病--肝细胞癌(HCC)的一个可行的蛋白质目标。然后他们从AlphaFold 蛋白质结构数据库中下载了 CDK20 的一个预测结构,并将其与市面上的生成化学平台 Chemistry42 结合起来,生成了近 10000 个小分子作为结合伙伴候选物。
通过各种可开发性的筛选,将总数缩减到 7 个分子,以便在湿式实验室中进行评估。在这 7 个候选分子中,有一个被发现具有很高的结合亲和力(Kd=9.2)。他们根据他们的热门分子的拟议作用机制重新进行了计算化学生成步骤,这使得结合亲和力进一步提高了 24 倍。
他们验证了他们的最佳候选分子在生化试验中降低了 CDK20 激酶的活性,而且与非癌细胞系 HEK 293 相比,还优先降低了肝癌细胞系的细胞增殖。
Ren 和她的团队展示了 AlphaFold 在其药物设计管道中的整合,具有显著的效率和速度。整个过程花了 30 天,在第一轮中只合成了 7 个化合物,另外在第二轮细化步骤中又合成了 6 个。这是一个多么令人振奋的时代。
发现隐蔽的结合袋
蛋白质与其他蛋白质和小分子相互作用的方式之一是通过结合。一个酶通常有一个与蛋白质活性部位相邻的结合袋,其中附着的分子改变了酶的行为。通过阻断底物(在同一口袋中结合)或改变蛋白质的形状(在其他地方结合)称为异生调节。
思考蛋白质结合的两种主要方式是锁和钥匙模型,以及手和手套模型。
- 锁和钥匙模型指出,蛋白质不灵活地与其底物或调节分子结合是高度具体的。
- 另一方面,手和手套模型承认,结合可以通过诱导配合来改变蛋白质的形状,就像一只手改变手套的形状一样。
在现实中,两种模型都是有用的,但都不完全正确。隐性口袋是潜在的结合点,没有出现在实验确定的蛋白质结构中,如冷冻电镜和X射线晶体学。蛋白质通常必须被纯化和结晶,这一过程可能会改变其原始结构。
最近,Meller 等人展示了使用 AlphaFold 来发现隐性口袋的方法。通过对一个蛋白质的多序列排列进行子采样,他们表明他们可以产生一个多样化的蛋白质结构。这些给定的蛋白质结构的变化可能更类似于体内蛋白质的动态和灵活状态,可作为分子动力学模拟的种子结构。
他们将他们的方法应用于 10 种已知具有隐性结合袋的蛋白质,并能够恢复其中 6 种蛋白质中存在的隐性结合袋。他们的数据集包括 5 个在 AlphaFold 训练时没有存入蛋白质数据库的结构,其中他们成功恢复了 3 个隐性结合袋。
Meller 等人的工作表明,使用 AlphaFold 的高质量结构模型来设计和发现以前被认为是无法通过常规手段进行药物治疗的蛋白质的潜力。他们还展示了 AlphaFold 的蛋白质结构与 GROMACS 的基于物理学的分子动力学模拟的互补应用潜力。
肽和蛋白质治疗剂的设计
在 CASP14 竞赛和 AlphaFold2 论文和代码发布之间,华盛顿大学的贝克实验室开发了 3 轨网络模型 RoseTTAFold。
由于采取了在硬件内存限制下工作的训练策略,研究人员发现 RoseTTAFold 可以直接生成多个相互作用的蛋白质的结构。
RoseTTAFold 的第一阶段是对裁剪过的序列进行训练,这些序列的特征随后被结构模块所使用。因此,RoseTTAFold 可以用来预测来自独立蛋白质的序列的复杂结构,跳过结构生成后的典型对接模拟步骤。
DeepMind 不甘落后,开发了一个专门针对蛋白质复合体训练的 AlphaFold 版本,被称为AlphaFold Multimer。蛋白质-蛋白质或肽-蛋白质的相互作用是开发治疗药物的另一种可行策略,一些团队已经将 RoseTTAFold 和 AlphaFold Multimer 如何提供帮助的演示放在一起。
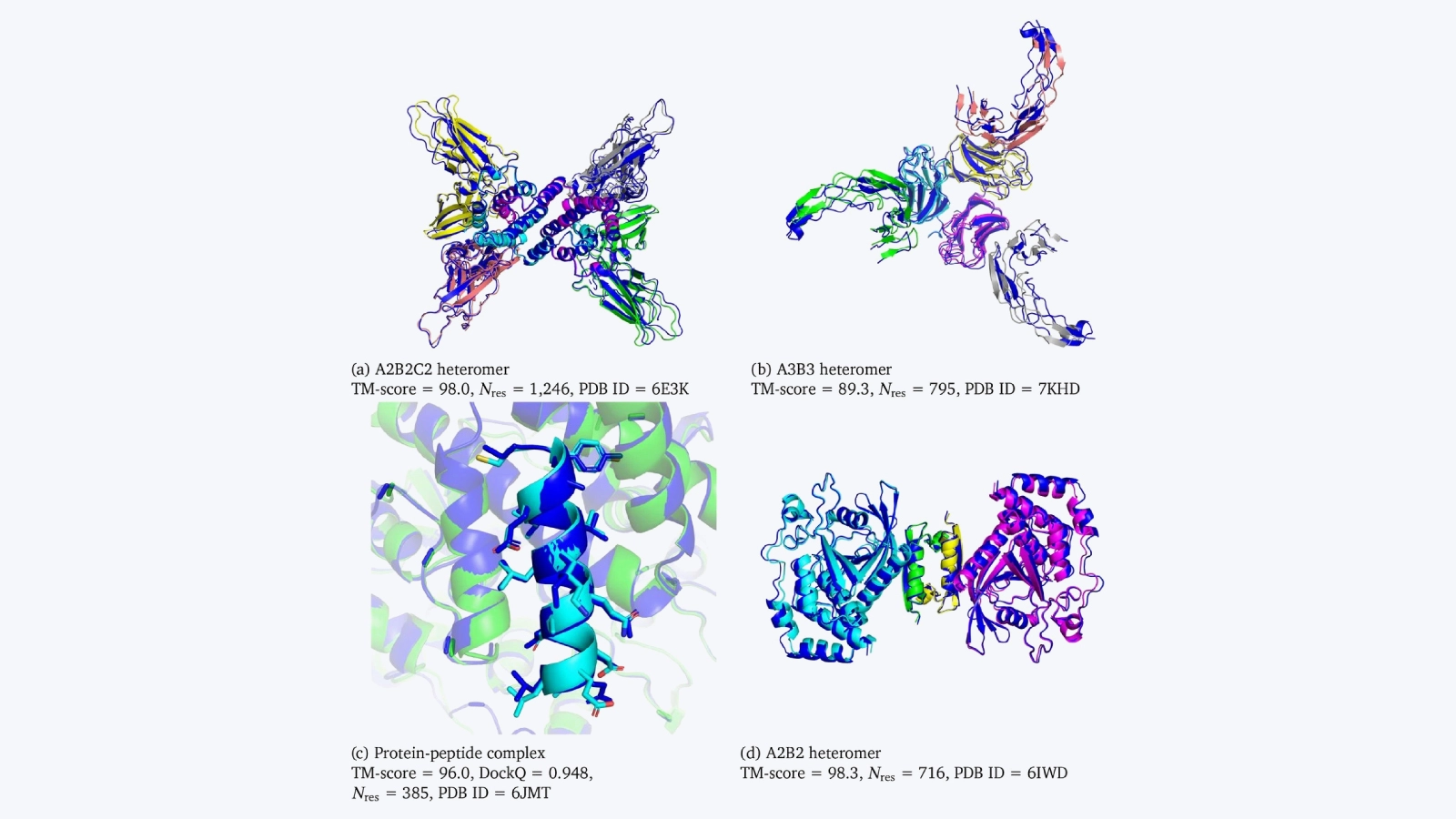
Åkhe 和 Wallner 表明,通过对 AlphaFold Multimer 的参数进行扰动,他们可以扩大模型探索构象空间的能力。他们报告了 AlphaFold 作为蛋白质-肽对接模拟的替代方案的改进能力,这是设计肽类药物的一个值得注意的优势。
为了使这类研究更加简化,AlphaPullown 是 Yu、Kosinsk i及其同事开发的一个 Python 软件包,用于使用 AlphaFold Multimer 对蛋白质-蛋白质相互作用进行高通量筛选。
除了成为改变疾病表现的有效目标外,蛋白质还是一种有希望的工程治疗底物。蛋白质药物并不新鲜,重组胰岛素(一种肽类药物)于1982年被美国食品和药物管理局批准,而著名的改变自身免疫性疾病的融合蛋白--依那西普,于1998年首次被批准。像 AlphaFold 和 RoseTTAFold 这样准确的折叠预测工具的出现为这些类型的治疗药物的计算设计带来了巨大的希望。
ABLooper, DeepAb, and IgFold: 预测抗体结合区域,受 AlphaFold 的启发
人类免疫系统大量使用抗体来特异性地与致病细胞或蛋白质结合,使其无法运作,并将其标记为 T 细胞和巨噬细胞破坏。
在过去的几十年里,外源性抗体已被用于对抗棘手的疾病,如自身免疫性疾病、癌症,甚至 SARS-CoV-2 病毒。最早被 FDA 批准的单克隆抗体之一,阿达木单抗(品牌名 Humira ),直到最近都是最畅销的药物。
单克隆抗体随后经历了大量的研究、开发和投资兴趣,有100多种已批准的抗体药物可用,并且每年都有更多的药物被考虑纳入监管审批。
进化已经在抗体中选择了一个方便的、分区的结构。高度可变的互补性决定区(CDR)在抗原结合片段(Fab)中提供目标特异性,而结晶片段(Fc)提供稳定性。这意味着蛋白质工程师在设计一个新的治疗性抗体时,可以专注于相对较短的氨基酸序列。
抗体的内在设计优势也伴随着一些自身的挑战。也就是说,AlphaFold 在预测 CDR 中的环状结构方面表现较差,而 CDR 对治疗效果至关重要。专门的抗体可变区域结构模型包括牛津大学 Charlotte Dean 实验室开发的 ABLooper,以及 Jeffrey Ruffolo、Jeffrey Gray 和约翰霍普金斯大学的同事受 AlphaFold 启发而开发的 IgFold 和 DeepAb。即使它的缺点也使研究人员能够采用 AlphaFold 的训练思想来开发更好的工具,以解决他们的具体焦点。
关于利用蛋白质结构预测模型进行药物开发的最终想法
自 CASP13 和 CASP14 以来,蛋白质结构预测的发展速度很快,为改善人类健康、理解和缓解疾病提供了重要的贡献工具。许多工作强调了人工智能和 ML 蛋白质结构预测对于开发新的治疗方法的有用性,从小分子到肽和蛋白质药物。未来几年有望成为治疗疑难病症的富有成效的时期,包括开发治疗 "不可药用 "的蛋白质目标。
AlphaFold 和公司有效应用的硬件和软件要求与深度学习以及更传统的生物信息学管道有一些相似之处。由深度学习(神经)构件组成,AlphaFold 使用了一个由 "进化体 "和结构模型组成的两阶段模型。RoseTTAFold 的神经组件在其3轨设计中大量使用了旋转和平移不变的 SE(3) 变换器模块,包含了一维、二维和三维表示。
RoseTTAFold 和 AlphaFold 在具有大量显存的现代 GPU 上表现最好,如 RTX 4090(24GB)或RTX 6000 Ada(48GB)用于推理。除了在 GPU 上运行良好的深度学习转化器外,这两种方法还利用了多序列排列。为给定的蛋白质建立多序列对齐需要快速搜索大型数据库,在预测结构时,搜索、聚类和对齐序列可能会占用大部分的计算时间。
有许多不同的方法来使用蛋白质折叠模型或获取其输出,其中许多是免费和公开的。对于为以前在 PDB 中没有实验结构的蛋白质目标开发小分子药物,你可能能够从 AlphaFold 蛋白质数据库中获得你所需要的一切。
另一方面,如果一个项目需要产生多个结构变体来探索一个特定蛋白质的可能构象空间,(例如发现可药用的隐性口袋),你就需要一个具有灵活性和控制力的实现方法,以便充分利用先进方法的潜力。需要更多的控制来探索具有多个结构变体的构象空间或设计蛋白质或肽类治疗的新序列的用户可能需要完全控制他们自己的硬件和软件。
对于支持 AlphaFold 和 RoseTTAFold 模型的交钥匙系统,并辅以对高性能分子动力学的可靠支持,请考虑联泰集群公司在生命科学工作流程方面的长期专业知识。我们提供工作站、服务器和集群解决方案,根据您的蛋白质结构工程需求进行定制和优化,同时满足任何其他科学计算要求。
相关贴子
-
 技术分享
技术分享什么是 OCP 3.0 NIC?
2025.07.25 30分钟阅读 -
 技术分享
技术分享GPU 如何加速高通量筛选研究
2025.09.26 39分钟阅读 -
 技术分享
技术分享YOLOv8 目标检测设置教程
2025.05.30 47分钟阅读






